2. Les microsatellites ou Polymorphisme de nombre d'unités de répétition
Sur le génome, il existe des séquences constituées d'unités répétées de 1 à 4 nucléotides. Ce sont les microsatellites. Les plus courants sont (A)n, (TC)n, (TAT)n et (GATA)n, les valeurs de n pouvant aller de quelques unités à plusieurs dizaines. On parle de séquences répétées en tandem ou SSR (Simple Sequence Repeats). L'intérêt de ces microsatellites réside dans leur polymorphisme. Celui-ci repose sur la variation du nombre d'unités de répétition, constituant le microsatellite.
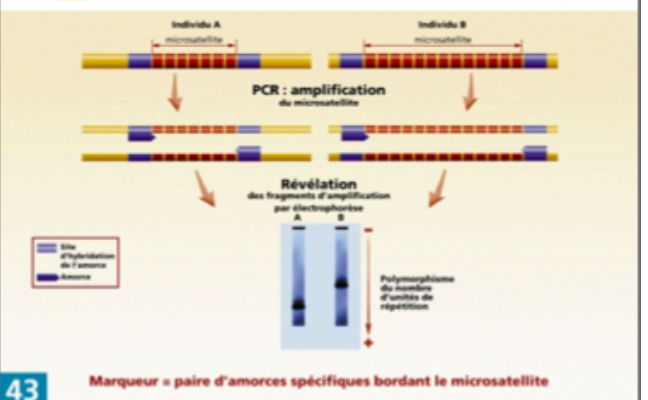
Les étapes de la technique
C'est la technique de PCR qui est utilisée pour révéler le polymorphisme des microsatellites. Une paire d'amorces spécifiques des bordures droite et gauche d'un microsatellite est utilisée pour amplifier le même microsatellite chez différents individus. En effet, chaque microsatellite est bordé par des séquences uniques qui lui sont propres. Les fragments d'amplification sont ensuite révélés par électrophorèse.
Sur la figure l’individu B, possédant plus d'unités de répétition que A, a un produit d'amplification qui migre plus lentement que A.
Création de marqueurs
C'est la paire d'amorces spécifiques des bordures droite et gauche du microsatellite qui constitue le marqueur.
Utilisation de la technique
C'est une technique qui nécessite une préparation préalable assez lourde. Il faut en effet connaître, synthétiser et tester les amorces bordant le microsatellite. En revanche, elle est simple d'utilisation car reposant simplement sur une PCR. Elle permet de développer de nombreux marqueurs, notamment sur le maïs ou le colza. Toutefois, elle n'est pas applicable à toutes les espèces, la tomate par exemple ne possède pas de polymorphisme pour les microsatellites.
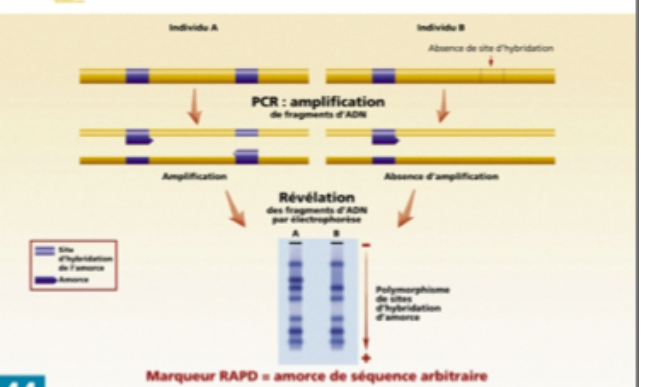
ADN polymorphe amplifié au hasard (Random Amplified Polymorphic DNA) RAPD
Cette technique consiste à réaliser une PCR en utilisant une amorce courte d'une dizaine de nucléotides, de séquence arbitraire. Cette amorce va s'hybrider au hasard dans le génome. Si deux sites d'hybridation sont proches et sur deux brins d'ADN, il y aura amplification, c'est le cas de l'individu A. En revanche, si ces deux sites sont trop éloignés, il ne peut y avoir amplification, cas de l'individu B. Ainsi, on observe la présence d'une bande sur le gel pour A et l'absence pour B de cette même bande.
Le polymorphisme révélé est un polymorphisme de sites d'hybridation d'amorce.
Les amorces constituent donc les marqueurs. Pour l'ensemble du génome, une dizaine de fragments sont en moyenne amplifiés puis séparés par électrophorèse.
Utilisation de la technique
Cette méthode ne nécessite pas de digestion par une enzyme de restriction, pas de transfert sur une membrane, pas de préparation de sonde radioactive. Elle est donc rapide et d'une faible technicité. Toutefois, sa reproductibilité est difficile à obtenir. En effet, l'amplification obtenue dépend beaucoup des conditions de réalisation de la PCR. Ainsi, les résultats sont difficilement reproductibles d'un laboratoire à un autre.
Elle est utilisée lors d'analyses préliminaires rapides, par exemple la mise en évidence de quelques marqueurs au voisinage d'un gène d'intérêt.
Polymorphisme de longueur des fragments d'amplification (Amplification Fragment Length Polymorphism)
Cette technique est fondée sur la mise en évidence conjointe de polymorphisme de site de restriction et de polymorphisme d'hybridation d'une amorce de séquence arbitraire.
Les étapes de la technique
L'ADN de la plante est soumis à une digestion par des enzymes de restriction. Les tailles des fragments obtenus sont dépendantes des enzymes utilisées.
Ensuite, il y a addition aux extrémités des fragments de restriction d'adaptateurs nucléotidiques spécifiques des enzymes de restriction utilisées. Ils sont de séquences connues.
Les fragments sont ensuite amplifiés par PCR. On utilise comme amorce un oligonucléotide complémentaire de la séquence de l'adaptateur, prolongé de quelques nucléotides arbitraires (de 1 à 3) appelés bases débordantes. Seuls sont amplifiés les fragments possédant les bases complémentaires de ces bases arbitraires. Il s'agit donc d'amorces sélectives permettant de réduire le nombre de fragments amplifiés à une centaine : sans ces séquences débordantes, il y aurait amplification de milliers de fragments. Les bandes sont visualisées par électrophorèse.
Création de marqueurs
C'est la combinaison enzyme de restriction/amorce qui permet de révéler le polymorphisme entre les individus. Celle-ci constitue le marqueur AFLP. Le locus mis en évidence dépend de la séquence du site de l'enzyme de restriction et des bases arbitraires. Il existe de très nombreuses combinaisons enzyme/amorce. En effet, on dispose d'une dizaine d'enzymes de restriction et il existe de très nombreuses amorces d'amplification par combinaisons de 3 bases débordantes.
Les marqueurs SNP Polymorphisme mononucléotidique : SNP (Single Nucleotide Polymorphisme)
Les récents progrès en matière d'analyse de séquences d'ADN et la mise au point de méthodologies à haut débit ont rendu possible l'identification et l'analyse de la variation nucléotidique à grande échelle.
Le marquage moléculaire par SNP permet de repérer les différences au niveau d'un nucléotide dans une séquence d'ADN. Cette technique consiste à hybrider sur l'ADN cible une sonde complémentaire portant une molécule fluorescente (le fluorophore). Chaque sonde est spécifique d'une séquence d'ADN donnée.
La deuxième étape consiste en une élongation par action de la Taq polymérase (PCR). Cette enzyme ajoute à l'extrémité des amorces des oligonucléotides présents dans le milieu de réaction et libère les fluorophores fixés sur les sondes.
Enfin, une visualisation par excitation et quantification du fluorophore, à une longueur d'onde qui lui est propre, est réalisée. Les individus A et B ne révèlent pas la même fluorescence, il ont donc un polymorphisme au niveau d'un nucléotide.
- Utilisation de la technique
Cette technologie qui permet d'éliminer entièrement les étapes de séparation de taille par électrophorèse présente un potentiel d'automatisation très supérieur aux technologies précédentes (RFLP, RAPD, AFLP, SSR...). Elle peut donc être réalisée à très haut débit. Le marquage SNP ou Snip permet d'obtenir des résultats précis pour différencier des allèles entre individus
