Les puces à ADN
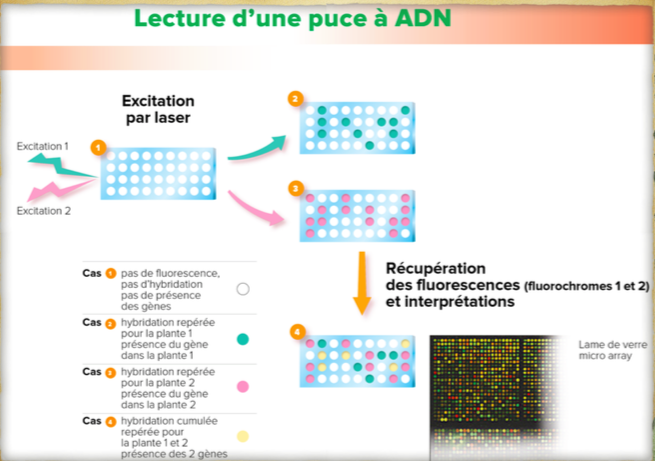
Les puces à ADN sont des images fonctionnelles du génome. Avec l’aide de plusieurs milliers de sondes, il est possible de comparer finement deux plantes. Après excitation laser des lames de verre sur lesquelles les échantillons des plantes ont été déposés, l’interprétation des couleurs de puits permet de déceler les différences génétiques de deux plantes.
Les applications
Les puces à ADN permettent d’étudier le transcriptome, c’est-à-dire l’expression des séquences codantes des gènes transcrits en ARN messagers (simple brin). C’est une image fonctionnel du génome, puisque les biopuces ou puces à ADN vont révéler les séquences codantes du génome effectivement exprimées.
La technique repose sur l’hybridation de deux fragments d’acides nucléiques complémentaires ou leur dissociation sous l’action de la température et de la concentration en sel du milieu.
Conception des puces à ADN
Une puce à ADN est un support rigide (verre ou nylon) de petite taille sur lequel sont fixés de courtes séquences d’ADN, appelées sondes qui correspondent à des fragments spécifiques (uniques) d’un seul gène. L’ensemble des gènes étudiés sont déposés sur la puce et chaque position est connue. Les sondes ne sont pas marquées.
Les cibles correspondent aux ARN messagers que l’on souhaite étudier. Pour cela, après extraction des ARN messagers, les chercheurs effectuent une transcription réverse pour obtenir des ADN complémentaires qui sont marqués avec des fluorochromes (ou par des marqueurs radioactifs).
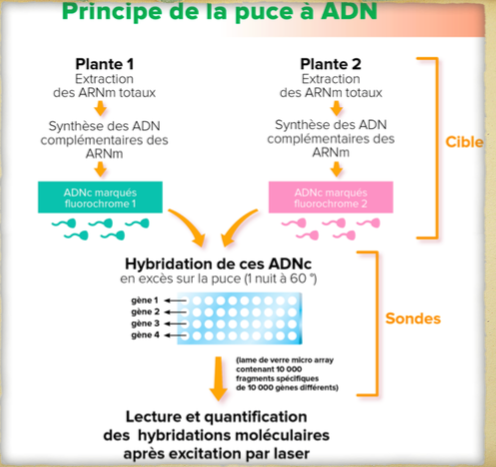
Interaction sonde/cible
Elle se fait en 12 heures à 60° C environ par interaction des deux chaines de séquences complémentaires qui correspond à une hybridation moléculaire. Si le nombre de sondes sur la puce est bien supérieur aux nombres de cibles (venant s’hybrider sur la puce)Utilisation des puces à ADN
Les puces à ADN permettent de regrouper des gènes ayant le même profil d’expression dans des conditions expérimentales particulières. Elles peuvent par exemple être un outil performant donc de nombreux gènes, car plusieurs milliers, voire plusieurs millions de substitutions de nucléotides peuvent être ainsi étudiées simultanément.
Les différentes puces à ADN
Les filtres à haute densité (macroarrays) sont des plaques en nylon (12X8 cm) qui permettent de quantifier la présence de 2400 gènes par marquage radioactif d’un échantillon (ARNm totaux à étudier).
Les lames de verre (microarrays) permettent de quantifier la présence de 10 000 gènes par marquage fluorescent avec deux conditions expérimentales par lame
Les puces à oligonucléotides (puce de 1,28cm x 1,28cm) contiennent 300 000 oligonucléotides (sondes) par lame et permettent de quantifier la présence d’ARNm grâce au marquage fluorescent de ces cibles issues d’une seule condition expérimentale.
L'empreinte génétique d'une plante
Les marqueurs moléculaires permettent d’établir l’empreinte génétique d’une plante.
Les puces à ADN permettent de visualiser et de mesurer très rapidement des milliers de différences d’expression au niveau d’un seul nucléotide entre de très nombreux gènes.
Applications
Cette identification peut intervenir à différents niveaux :
• L’identification variétale. Par ces techniques, il devient possible de distinguer les lignées et de reconnaître les parents d’hybrides.
• Le contrôle de la pureté variétale. Il s’agit de détecter des impuretés variétales de lots de semences, ou de tester l’homogénéité
d’une population à n’importe quel stade d’un schéma de sélection. • La protection variétale. Ceci fait partie intégrante de l’identification
variétale. Il devient possible de détecter les contrefaçons ou copies de génotypes. Ainsi, pour des questions de droits liés aux obtentions végétales, il a été introduit la notion de variété essentiellement dérivée (VED) : c’est une variété dérivée d’une variété originale par modification de quelques zones chromosomiques réduites.

Les outils complémentaires de la génomique
Grâce à la miniaturisation et à l’automatisation des outils de la génomique, le coût et le temps nécessaires à la caractérisation des fragments d’ADN ou de génomes sont en forte diminution d’année en année. Mais pour obtenir des connaissances biologiques à partir des données de génomique, il est nécessaire de réaliser des analyses informatiques complexes ainsi que de très nombreuses mesures et observations sur les plantes. Des laboratoires spécialisés appelés plateformes peuvent prendre en charge ces différents aspects.
Les plateformes bio-informatique
• Elles intègrent les données produites par les appareils de séquençage,
• Elles assurent l’analyse informatique et statistique des données en produisant des cartes génomiques ou des bases de données
et elles guident la recherche vers les gènes actifs ou de régulation les plus intéressants.
Les plateformes de phénotypage
Le phénotypage est l’observation et la mesure du phénotype, c’est-à-dire du caractère d’intérêt. Pour étudier les gènes qui contrôlent un caractère, par exemple la tolérance au stress hydrique, il est nécessaire de mesurer très précisément ce caractère sur de nombreuses plantes grâce à une collection de mutants. En inactivant un gène et en observant les conséquences physiologiques, il est possible de corréler un gène et sa fonction. Les plateformes de phénotypage permettent de réaliser ces mesures à haut débit sur des centaines de plantes différentes, grâce à des robots qui mesurent, pèsent, et photographient les plantes automatiquement et en font des analyses d’image.
Les plateformes de transcriptomique, de protéomique et de métabolomique
Les technologies « omiques » sont des techniques à haut débit qui permettent de générer de grandes quantités de données à des niveaux biologiques multiples : du séquençage des gènes à l’expression des protéines et des structures métaboliques. Ces données peuvent couvrir tous les mécanismes impliqués dans les variations qui se produisent dans les cellules et qui influent la vie de l’organisme.
Elles comprennent la transcriptomique (expression des gènes et de leur régulation), (voir chapitre Les marqueurs moléculaires), la protéomique (étude des protéines), la métabolomique (étude des métabolites cellulaires produits lors des réactions cellulaires) ou encore l’épigénomique (étude des régulations, parfois héritables, de l’expression des gènes qui ne s’accompagnent pas de changement de séquence de l’ADN, via la méthylation ou autres modifications chimiques, ou les ARN non codants).
Des études scientifiques sont déjà parues sur Arabidopsis, plante modèle de laboratoire mais aussi sur les céréales, la pomme de terre ou la tomate. Elles peuvent compléter les études réalisées pour évaluer l’équivalence en substance entre deux variétés.

